Effects of circ-0005273 targeting miR-1200 on the proliferation and apoptosis of esophageal cancer Eca109 cells
-
摘要:目的 探讨circ-0005273对食管癌细胞增殖和凋亡的影响及可能机制。方法 收集41例食管癌患者的癌组织及癌旁组织,采用实时荧光定量聚合酶链式反应(RT-qPCR)检测组织中circ-0005273和miR-1200表达。体外培养食管癌Eca109细胞,分别转染circ-0005273小干扰RNA、miR-1200模拟物,或共转染circ-0005273小干扰RNA与miR-1200抑制剂。采用CCK-8法和克隆形成实验检测细胞增殖;流式细胞术检测细胞凋亡;蛋白印迹法检测细胞中cleaved-caspase9、cleaved-caspase3蛋白表达;双荧光素酶报告基因实验验证miR-1200与circ-0005273调控关系。结果 食管癌组织中circ-0005273的表达量高于癌旁组织,而miR-1200的表达量低于癌旁组织,差异均有统计学意义(P<0.05)。干扰circ-0005273表达或过表达miR-1200后,Eca109细胞光密度(OD)值和克隆形成数降低,凋亡率和cleavde-caspase9、cleavde-caspase3蛋白的表达量增加,差异均有统计学意义(P<0.05)。circ-0005273靶向负调控miR-1200,干扰miR-1200可逆转干扰circ-0005273对Eca109细胞增殖和凋亡的影响。结论 circ-0005273在食管癌组织中呈高表达,干扰circ-0005273可能通过靶向负调控miR-1200阻碍了食管癌细胞增殖,并加剧了细胞凋亡。
-
关键词:
- 食管癌 /
- circ-0005273 /
- miR-1200 /
- 细胞增殖 /
- 凋亡
Abstract:Objective To investigate the effects of circ-0005273 on the proliferation and apoptosis of esophageal cancer cells and its possible mechanism.Methods The cancer tissues and adjacent tissues of 41 patients with esophageal cancer were collected, and the expression of circ-0005273 and miR-1200 in the tissues were detected by real-time fluorescence quantitative polymerase chain reaction (RT-qPCR). Esophageal cancer Eca109 cells were cultured in vitro, and were respectively transfected with circ-0005273 small interfering RNA (si-circ-0005273) or miR-1200 mimic, or co-transfected with circ-0005273 small interfering RNA and miR-1200 inhibitor. CCK-8 method and clone formation experiment were used to detect cell proliferation, flow cytometry was used to detect cell apoptosis, the western blotting was used to detect the protein expression of cleaved-caspase9 and cleaved-caspase3 in cells, and the dual luciferase reporter gene experiment was used to verify the regulatory relationship between miR-1200 and circ-0005273.Results The expression of circ-0005273 in esophageal cancer tissues was significantly higher than that in adjacent tissues, but the expression of miR-1200 was significantly lower than that in adjacent tissues (P < 0.05). After disturbing circ-0005273 or over-expressing miR-1200, the optical density (OD) value and clone formation number of Eca109 cells significantly decreased (P < 0.05), but the apoptosis rate and the protein expression of cleavde-caspase9 and cleavde-caspase3 increased significantly (P < 0.05). Circ-0005273 was able to target and negatively regulate miR-1200, and disturbing miR-1200 reversed the effect of disturbing circ-0005273 on the proliferation and apoptosis of Eca109 cells.Conclusion Circ-0005273 is highly expressed in esophageal cancer tissues. Disturbing circ-0005273 may hinder the proliferation of esophageal cancer cells and aggravate cell apoptosis by targeting and negatively regulating miR-1200.-
Keywords:
- esophageal cancer /
- circ-0005273 /
- miR-1200 /
- cell proliferation /
- apoptosis
-
年龄相关性白内障(ARC)为世界首位致盲性眼病,是一种衰老相关的慢性病,其发病率随年龄增长而增加[1-2]。目前,对ARC发生发展的具体分子机制仍然缺乏较全面的了解。长链非编码RNA(LncRNA)是长度大于200个核苷酸的非编码RNA, 可在基因的转录、转录后和翻译后等水平调控其表达,在多种生理学和病理学过程中发挥重要作用[3]。研究[4]发现大量LncRNA参与细胞衰老及相关信号通路的调控,影响关键细胞周期过程,如细胞增殖、分化、静止、衰老等。这类衰老相关LncRNA通过表观遗传调控、细胞增殖、端粒稳定性、蛋白质内稳态、细胞间通讯、干细胞池等调控衰老[5]。本研究探讨衰老相关LncRNA PINT对ARC发生发展的影响,现报告如下。
1. 材料与方法
1.1 研究对象
病例组(ARC组):在扬州大学附属医院眼科被诊断为ARC、接受白内障小切口手法碎核白内障手术患者。收集中度皮质性ARC、重度皮质性ARC、中度核性ARC、重度核性ARC各10眼。纳入标准: 患者年龄50~79岁; 最佳矫正视力低于0.5; 排除标准: 先天性、糖尿病性、外伤性、并发性、药物及中毒性白内障等患者。所有参加研究的患者均无高血压病、糖尿病、自身免疫性疾病及其他氧化损伤性相关疾病。
对照组: ①在扬州大学附属医院眼科被诊断为玻璃体视网膜疾病(如特发性黄斑前膜、特发性黄斑裂孔等),因年龄和手术等因素需要在玻璃体切除手术中同时行透明晶状体摘除者,共10眼。②扬州市红十字眼库捐献的新鲜眼球。纳入标准[6]: 年龄50~79岁; 晶状体透明。排除标准: 高血压病、糖尿病、自身免疫病及其他氧化损伤性相关疾病者。研究对象签署知情同意书。③人晶状体上皮细胞株SRA01/04(Thermo Fisher Scientific公司)。上述研究方案严格遵循赫尔辛基宣言,且经扬州大学医学院伦理委员会批准。
1.2 RNA提取、逆转录和实时荧光定量PCR
使用TRIzol试剂(Invitrogen, Carlsbad, CA, United States)从ARC和对照组细胞中提取总RNA, 然后使用PrimeScript RT Master Mix(Takara, 中国大连)、Power SYBR Green PCR Master Mix(Applied Biosystems, Irvine, CA, United States)在ABI 7500实时PCR系统上进行实时PCR分析。选用actin beta作为内参,每个实验重复3次。
1.3 细胞培养和处理
人晶状体上皮细胞株SRA01/04起源于人晶状体上皮细胞,将其培养在DMEM中,含有10%胎牛血清(FBS), 青霉素(100单位/mL)和链霉素(100 μg/mL)。所有细胞在37 ℃, 95%空气和5%二氧化碳的加湿培养箱中培养。氧化损伤组:当生长至80%~90%时,将细胞暴露于过氧化氢(H2O2)(Sigma-Aldrich Corp., St Louis, MO, USA)中,进行氧化损伤。将细胞在含2%FBS的DMEM中孵育过夜,然后在无血清DMEM中孵育30 min。用H2O2(100和200μmol/L)处理24 h后,收集细胞进行不同的测定。衰老组:当细胞生长至80%~90%时,将细胞暴露于小剂量H2O2进行衰老处理。将细胞在含2% FBS的DMEM中孵育过夜,然后在无血清DMEM中孵育30 min, 并用50 μmol/L H2O2培养至7、14、21 d。实验结束时,收取所有细胞并用于不同的检测。平行培养的对照细胞在类似的培养基中生长,不进行处理,作为所有实验的对照。
1.4 质粒构建、慢病毒包装和转染
使用pCDNA3.1载体(Genechem Technology Co., Shanghai, China)。过表达LINC-PINT: 通过PCR生成LINC-PINT序列并克隆到pcDNA3.1载体中。慢病毒包装:将pcDNA、pcDNA-LINC-PINT、si-NC、si-LINC-PINT分别转染进人晶状体上皮细胞株SRA01/04中,转染6 h后,更换新鲜培养液继续培养48 h, 检测转染效率。转染成功后,用于后续实验。
1.5 细胞增殖能力测定
利用CCK8检测试剂盒(Dojindo Laboratories, Kumamoto, Japan)检测各组细胞的生长增殖情况,分别向处理或未处理的细胞中加入10 μL CCK8试剂, 37 ℃避光孵育3 h, 酶标仪检测450 nm处的吸光度(OD值),实验重复3次。
1.6 抗氧化能力检测
总抗氧化能力(T-AOC)检测试剂盒(FRAP法)测定总抗氧化水平。在酸性环境下,还原铁-三吡啶三吖嗪(Fe3+-TPTZ)产生亚铁离子(Fe2+)形式的能力反映了总抗氧化能力。收集100~200万个细胞,加入1.0 mL预冷的提取液,超声充分破碎细胞并释放其中的抗氧化物, 4 ℃、10 000次/min, 离心5 min, 取上清液。样品的抗氧化能力以达到同样吸光度变化值所需的标准液离子浓度表示。
1.7 染色质免疫共沉淀(ChIP)技术
ChIP测定: 使用EZ-Magna ChIP A/G试剂盒(Millipore), 应用抗EZH2(Active Motif)和抗H3K27me3(Active Motif)。将SRA01/04细胞与1%甲醛交联8 min, 然后匀浆。使用Branson Digital Sonifier(Model 450; Branson Ultrasonics Corporation, CT, USA)的微尖探针将匀浆在2级超声处理4次,每次15 s, 每次脉冲之间在冰上间隔40 s, 产生长度为200~800碱基对(bp)的DNA片段。ChIP分析:将等量处理过的染色质添加到含有针对目标蛋白的固定化抗体或阴性对照正常兔IgG抗体的微孔中。在65 ℃温育90 min以逆转交联并洗脱DNA后,使用快速旋转柱进行DNA纯化,然后进行实时PCR检测。
1.8 统计分析
采用IBM SPSS Statistics 20.0软件进行数据分析,所有结果数据以平均值±标准差表示。2组之间的差异采用非配对的Student′s t检验或方差分析进行比较。P < 0.05为差异具有统计学意义。
2. 结果
2.1 LINC-PINT在ARC中的表达
使用高通量RNA序列分析、确定ARC组和对照组晶状体上皮细胞之间的差异表达。ARC组LINC-PINT的表达水平高于对照组,差异有统计学意义(P < 0.05)。LINC-PINT的表达水平与ARC分级呈正相关。见图 1。
2.2 衰老相关LncRNA与氧化损伤/衰老的关系
在晶状体上皮细胞中,氧化损伤组和衰老组中LINC-PINT表达水平高于对照组,差异有统计学意义(P < 0.05), 且分别呈现为剂量依赖性或时间依赖性的正相关,见图 2、图 3。
2.3 建立过表达/敲低模型
将pcDNA、pcDNA-LINC-PINT、si-NC、si-LINC-PINT分别转染进人晶状体上皮细胞SRA01/04中,通过实时荧光定量PCR检测转染前后细胞中相关基因的表达量变化,发现转入pcDNA-LINC-PINT后,靶基因CCNA2、CDK1、PCNA表达减少; 转入si-LINC-PINT后,靶基因CCNA2、CDK1、PCNA表达增加,见图 4。同时, LINC-PINT改变抗氧化损伤能力,见图 5。CCK8显示在LINC-PINT过表达的细胞中细胞增殖被抑制,见图 6。
2.4 衰老相关LncRNA与转录因子的调控机制
在模拟物和LINC-PINT过表达的细胞中进行ChIP, 测定表观遗传修饰物与LINC-PINT靶基因的启动子区域之间的相互作用。LINC-PINT与EZH2相互作用。LINC-PINT将EZH2招募到CDK1、CCNA2和PCNA基因的启动子上,导致H3K27三甲基化和靶基因的表观遗传学沉默。见图 7。
3. 讨论
LncRNA PINT是由p53诱导的核LncRNA, 直接与PRC2相互作用,且是PRC2特异靶基因中的组蛋白H3K27甲基化所必需的。研究[7]表明, LINC-PINT的功能依赖于PRC2的表达水平,并通过与PRC2相互作用调控转化生长因子-β(TGF-β)、丝裂原活化蛋白激酶(MAPK)和p53细胞通路中的蛋白质表达水平,而这些通路与氧化损伤、衰老和衰老相关性疾病有关。p53及其下游p21蛋白是重要的细胞周期相关蛋白,广泛参与了细胞的增殖、分化、凋亡等多种生理及病理过程。研究[4, 8]表明, LncRNA是p53/p21信号通路的重要组成部分,而调控表观遗传学变异的LncRNA PINT在p53/p21信号通路中扮演着重要的角色。p53能通过诱导LncRNA PINT的表达,与PRC2相互作用,从而终止基因转录,调控p53信号通路中蛋白质的表达水平,调控细胞周期进程,从而影响机体的衰老以及衰老相关疾病的发生发展。
ARC与表观遗传因素密不可分。研究[9-10]报道, LncRNA与许多眼病相关,如青光眼、后发性白内障、糖尿病性视网膜病变、增生性玻璃体视网膜病变、眼部肿瘤等。其中有报道[11]表明LncRNA PAUPAR通过调控组蛋白H3K4的去甲基化进而抑制葡萄膜黑色素瘤的发生。作者的前期研究[12-13]提示,在人晶状体上皮细胞株SRA01/04中,高浓度短时间的氧化损伤可诱导氧化损伤模型,而低浓度长时间的氧化损伤可诱导衰老模型。ARC的衰老发病机制与氧化损伤机制紧密相扣,氧化损伤相关的表观遗传学改变与ARC密切相关[14]。因此,衰老相关LncRNA可能通过参与表观遗传学调控影响细胞衰老,进而引起ARC。
本研究初步阐明了LncRNA PINT可能通过p53/p21通路调控相关基因的表观遗传改变,参与ARC的发生发展,揭示了衰老相关LncRNA在ARC发病机制中起到重要作用,可作为ARC防治的新靶点。本研究从表观遗传学水平探讨ARC发病机制,为ARC防治提供新思路,有助于研发抗ARC药物,为进一步深入了解白内障的发病机理提供了理论基础和方向。
-
表 1 干扰circ-0005273表达对食管癌Eca109细胞增殖的影响(x±s)(n=9)
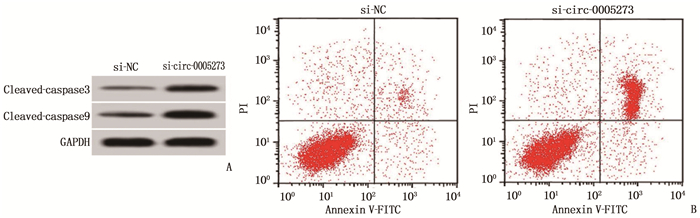
组别 circ-0005273 OD450 nm值 克隆形成数/个 si-NC组 1.00±0 0.81±0.06 92.24±7.81 si-circ-0005273组 0.33±0.03* 0.35±0.03* 41.03±4.08* 与si-NC组比较, * P<0.05。 表 2 干扰circ-0005273表达对食管癌Eca109细胞凋亡的影响(x±s)(n=9)
组别 凋亡率/% cleavde-caspase3蛋白 cleavde-caspase9蛋白 si-NC组 6.11±0.56 0.24±0.02 0.31±0.03 si-circ-0005273组 22.86±2.53* 0.65±0.04* 0.75±0.05* 与si-NC组比较, * P<0.05。 表 3 过表达miR-1200对食管癌Eca109细胞增殖和凋亡的影响(x±s)(n=9)
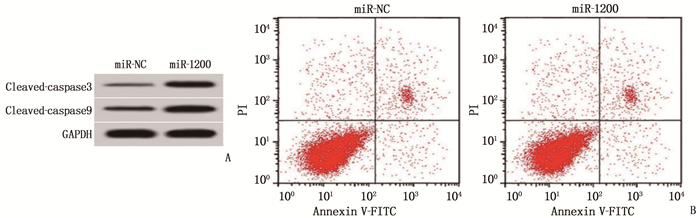
组别 miR-1200 OD450 nm值 克隆形成数/个 凋亡率/% cleaved-caspase3蛋白 cleaved-caspase9蛋白 miR-NC组 1.00±0 0.83±0.07 94.51±7.93 6.49±0.53 0.23±0.02 0.29±0.02 miR-1200组 3.12±0.25* 0.42±0.05* 50.76±5.99* 18.97±1.62* 0.61±0.05* 0.69±0.05* 与miR-NC组比较, * P<0.05。 表 4 干扰miR-1200逆转干扰circ-0005273对食管癌Eca109细胞增殖和凋亡的作用(x±s)(n=9)
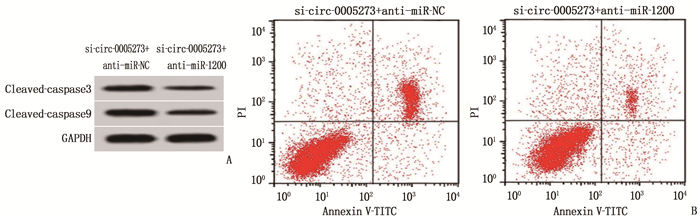
组别 miR-1200 OD450 nm值 克隆形成数/个 凋亡率/% cleaved-caspase3蛋白 cleaved-caspase9蛋白 si-circ-0005273+anti-miR-NC组 1.00±0 0.34±0.03 40.33±4.15 23.86±2.66 0.67±0.05 0.76±0.06 si-circ-0005273+anti-miR-1200组 0.39±0.04* 0.71±0.05* 83.48±6.56* 12.11±1.21* 0.32±0.03* 0.41±0.04* 与si-circ-0005273+anti-miR-NC组比较, * P<0.05。 -
[1] MENG L, LIU S, DING P, et al. Circular RNA ciRS-7 inhibits autophagy of ESCC cells by functioning as miR-1299 sponge to target EGFR signaling[J]. J Cell Biochem, 2020, 121(2): 1039-1049. doi: 10.1002/jcb.29339
[2] WANG H L, WANG H R, LIANG Y, et al. Hsa_circ_0006571 promotes spinal metastasis through sponging microRNA-138 to regulate sirtuin 1 expression in lung adenocarcinoma[J]. Transl Lung Cancer Res, 2020, 9(6): 2411-2427. doi: 10.21037/tlcr-20-1250
[3] ZHANG C C, LI Y, FENG X Z, et al. Circular RNA circ_0001287 inhibits the proliferation, metastasis, and radiosensitivity of non-small cell lung cancer cells by sponging microRNA miR-21 and up-regulating phosphatase and tensin homolog expression[J]. Bioengineered, 2021, 12(1): 414-425. doi: 10.1080/21655979.2021.1872191
[4] WEN Y, LI B, HE M, et al. circHIPK3 promotes proliferation and migration and invasion via regulation of miR-637/HDAC4 signaling in osteosarcoma cells[J]. Oncol Rep, 2021, 45(1): 169-179. http://www.ncbi.nlm.nih.gov/pubmed/33416147
[5] WANG X H, JI C L, HU J S, et al. Hsa_circ_0005273 facilitates breast cancer tumorigenesis by regulating YAP1-hippo signaling pathway[J]. J Exp Clin Cancer Res, 2021, 40(1): 29. doi: 10.1186/s13046-021-01830-z
[6] HOU Y S, LI X. Circ_0005273 induces the aggravation of pancreatic cancer by targeting KLF12[J]. Eur Rev Med Pharmacol Sci, 2020, 24(22): 11578-11586. https://pubmed.ncbi.nlm.nih.gov/33275224/
[7] YANG H, LI X, MENG Q, et al. CircPTK2(hsa_circ_0005273) as a novel therapeutic target for metastatic colorectal cancer[J]. Mol Cancer, 2020, 19(1): 13. doi: 10.1186/s12943-020-1139-3
[8] ZHANG W, ZHANG H, ZHAO X. circ_0005273 promotes thyroid carcinoma progression by SOX2 expression[J]. Endocr Relat Cancer, 2020, 27(1): 11-21. doi: 10.1530/ERC-19-0381
[9] PAN B, ZHAO M, WANG N, et al. LncRNA RGMB-AS1 promotes glioma growth and invasion through miR-1200/HOXB2 axis[J]. Onco Targets Ther, 2019, 12: 10107-10114. doi: 10.2147/OTT.S230098
[10] 刘明建. 肿瘤坏死因子受体相关蛋白1的表达与食管癌临床病理特征及预后的关系探讨[J]. 实用临床医药杂志, 2019, 23(6): 110-112. doi: 10.7619/jcmp.201906029 [11] WANG J, ZHU W, TAO G, et al. Circular RNA circ-LRP6 facilitates Myc-driven tumorigenesis in esophageal squamous cell cancer[J]. Bioengineered, 2020, 11(1): 932-938. doi: 10.1080/21655979.2020.1809922
[12] SHI Y, FANG N, LI Y, et al. Circular RNA LPAR3 sponges microRNA-198 to facilitate esophageal cancer migration, invasion, and metastasis[J]. Cancer Sci, 2020, 111(8): 2824-2836. doi: 10.1111/cas.14511
[13] ZOU F W, YANG S Z, LI W Y, et al. circRNA_001275 upregulates Wnt7a expression by competitively sponging miR-370-3p to promote cisplatin resistance in esophageal cancer[J]. Int J Oncol, 2020, 57(1): 151-160. http://www.ingentaconnect.com/content/sp/ijo/2020/00000057/00000001/art00012
[14] LI X Y, YANG B, SUN Y, et al. Effect of hsa_circ_RNA0023397 regulation of miR-106b expression on proliferation and apoptosis of esophageal cancer cells[J]. Eur Rev Med Pharmacol Sci, 2020, 24(17): 8855-8862. https://www.europeanreview.org/article/22825
[15] CHEN Z, YAO N, GU H, et al. Circular RNA_LARP4 sponges miR-1323 and hampers progression of esophageal squamous cell carcinoma through modulating PTEN/PI3K/AKT pathway[J]. Dig Dis Sci, 2020, 65(8): 2272-2283. doi: 10.1007/s10620-019-05973-0
[16] XING Y, ZHA W J, LI X M, et al. Circular RNA circ-Foxo3 inhibits esophageal squamous cell cancer progression via the miR-23a/PTEN axis[J]. J Cell Biochem, 2020, 121(3): 2595-2605. doi: 10.1002/jcb.29481
[17] 王晓庚, 刘林, 左健, 等. 沉默FoxM1通过促进线粒体释放细胞色素C诱导口腔鳞癌细胞凋亡[J]. 中国病理生理杂志, 2019, 35(3): 430-435. doi: 10.3969/j.issn.1000-4718.2019.03.008 [18] 余鑫, 米智华, 高巨. 自噬在机械通气致大鼠脑组织神经元凋亡中的作用[J]. 实用临床医药杂志, 2019, 23(8): 12-15, 21. doi: 10.7619/jcmp.201908003 [19] LIU Z, HU G, ZHAO Y, et al. Silence of cZNF292 suppresses the growth, migration, and invasion of human esophageal cancer Eca-109 cells via upregulating miR-206[J]. J Cell Biochem, 2020, 121(3): 2354-2362. doi: 10.1002/jcb.29458
[20] LI S, PEI Y, WANG W, et al. Circular RNA 0001785 regulates the pathogenesis of osteosarcoma as a CeRNA by sponging miR-1200 to upregulate HOXB2[J]. Cell Cycle, 2019, 18(11): 1281-1291. doi: 10.1080/15384101.2019.1618127
[21] ZHANG Z, YU X, ZHOU B, et al. Circular RNA circ_0026359 enhances cisplatin resistance in gastric cancer via targeting miR-1200/POLD4 pathway[J]. Biomed Res Int, 2020, 2020: 5103272. https://pubmed.ncbi.nlm.nih.gov/32855967/





 下载:
下载:







 苏公网安备 32100302010246号
苏公网安备 32100302010246号
