Molecular mechanism of luteolin in treatment of cervical cancer based on network pharmacology and molecular docking technology
-
摘要:目的
基于网络药理学与分子对接技术探讨木犀草素治疗宫颈癌的分子机制。
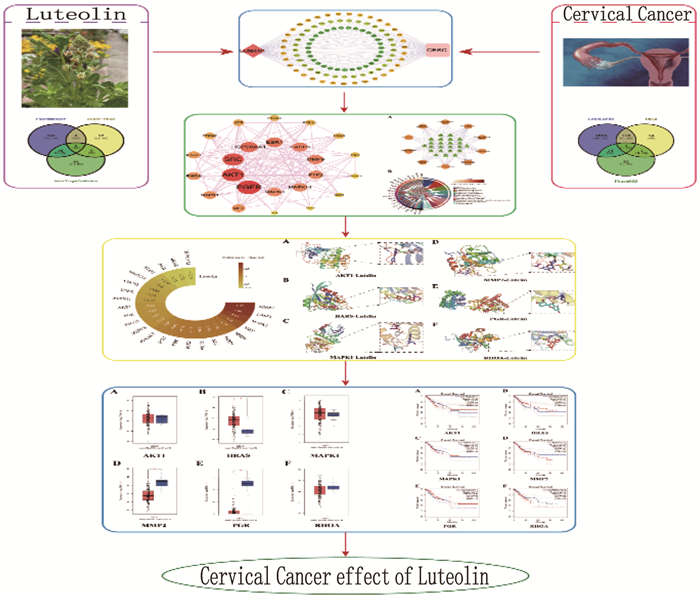
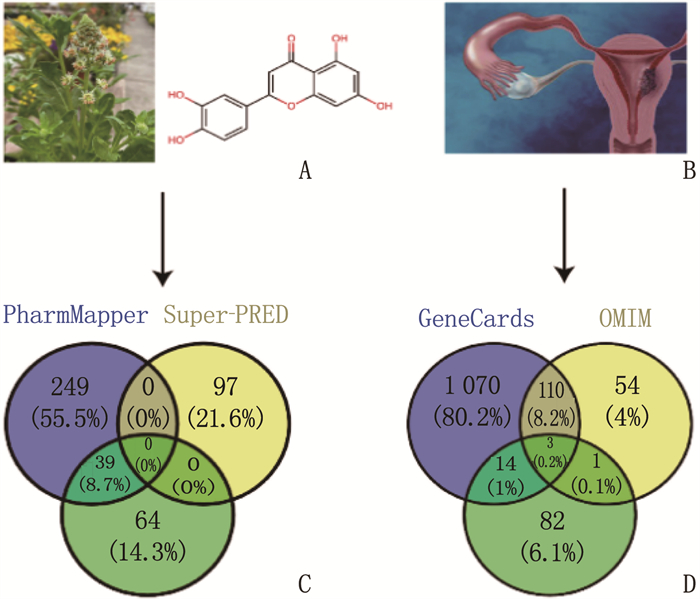
方法运用中药系统药理学数据库与分析平台(TCMSP)对木犀草素进行类药性分析。分别在PharmMapper、Super-PRED和Swiss Target Prediction数据库中获取木犀草素作用靶点。运用GeneCards、OMIM和PharmGKB数据库获取宫颈癌相关靶点。通过EVenn获得木犀草素与宫颈癌的交集靶点, 并使用Cytoscape3.8.1构建“木犀草素-交集靶点-宫颈癌”网络图。利用STRIING数据库对交集靶点进行蛋白质互作(PPI)网络分析,筛选核心靶点。采用David数据库对靶点进行基因本体论(GO)基因功能分析、京都基因和基因组百科全书(KEGG)信号通路富集分析。利用PyMoL 2.6.0、AutoDockTool 1.5.7和OpenBabel 2.4.1软件开展核心靶点与木犀草素的分子对接。将核心靶点在GEPIA数据库中进行生存分析及泛癌分析。
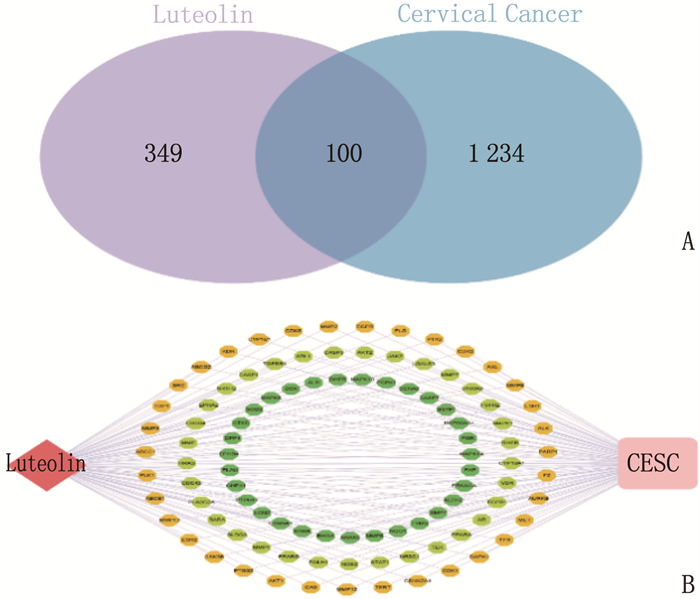
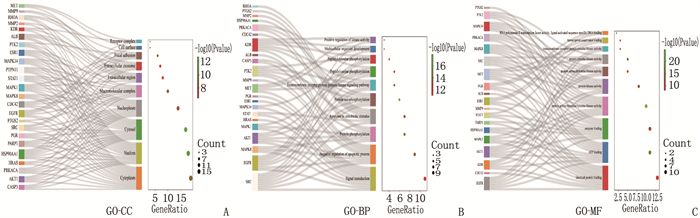
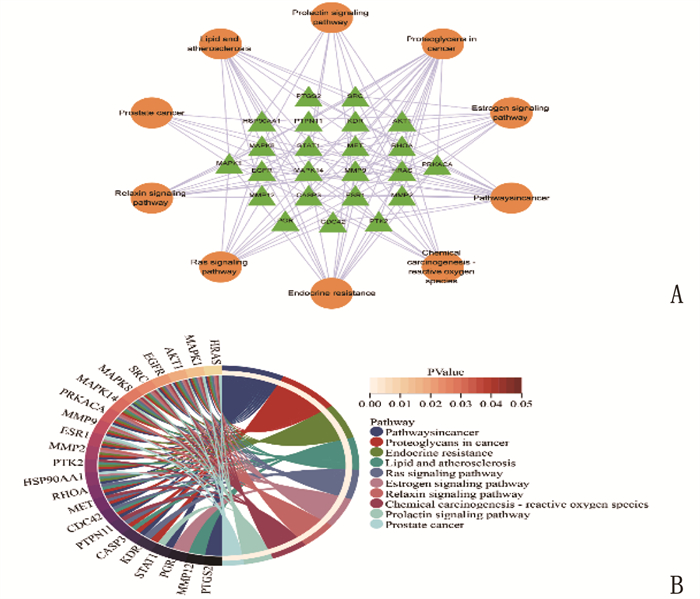
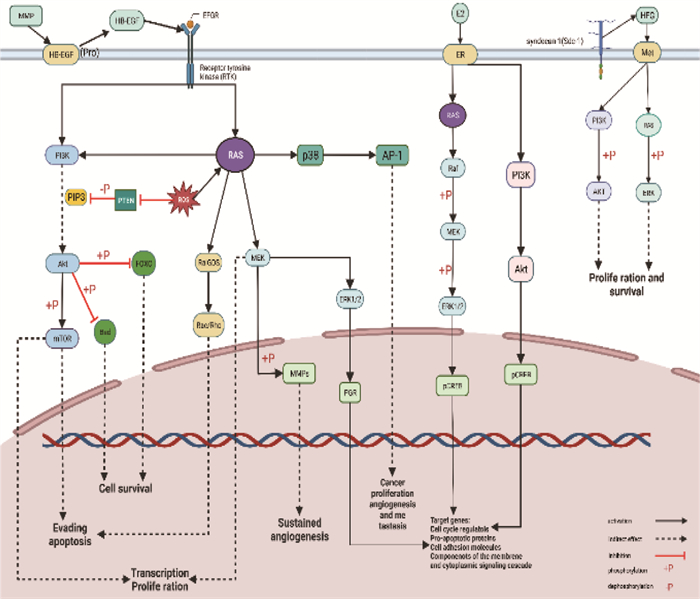
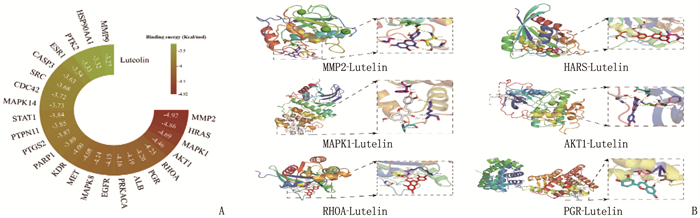
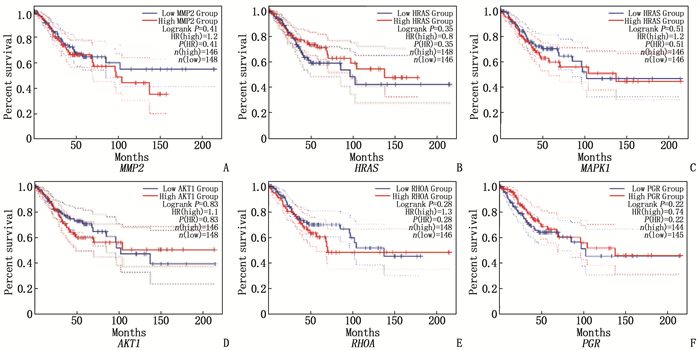
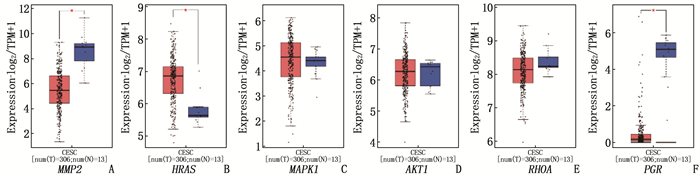
结果获得木犀草素靶点449个、宫颈癌相关靶点1 334个; 木犀草素与宫颈癌交集靶点100个,其中核心靶点有24个,包括MMP2、HRAS、MAPK1、AKT1、RHOA和PGR等。GO和KEGG富集分析发现交集靶点参与生物过程455条,细胞组分70条,分子功能119条和143条KEGG信号通路。分子对接发现MMP2与木犀草素结合较好。宫颈癌患者生存曲线显示RHOA、MAPK1、MMP2和AKT1基因风险比率>1, HRAS和PGR的风险比率 < 1。泛癌分析显示HRAS和MAPK1在宫颈癌中高表达,并且HRAS有显著表达差异。
结论木犀草素通过多靶点、多途径的作用方式治疗宫颈癌。
Abstract:ObjectiveTo explore the molecular mechanism of luteolin in the treatment of cervical cancer based on network pharmacology and molecular docking technology.
MethodsThe drug-like properties of luteolin were analyzed by the Traditional Chinese Medicine Systems Pharmacology Database and Analysis Platform (TCMSP). The targets of luteolin were obtained from PharmMapper, Super-PRED, and Swiss Target Prediction databases. The targets related to cervical cancer were acquired from GeneCards, OMIM, and PharmGKB databases. The intersection targets of luteolin and cervical cancer were obtained through EVenn, and the "luteolin-intersection targets-cervical cancer" network diagram was constructed by Cytoscape 3.8.1. The STRING database was used to analyze the protein-protein interaction (PPI) network of intersection targets and screen the core targets. The Database for Annotation, Visualization and Integrated Discovery (David) was used to conduct Gene Ontology (GO) gene function analysis and Kyoto Encyclopedia of Genes and Genomes (KEGG) signaling pathway enrichment analysis of the targets. PyMoL 2.6.0, AutoDockTool 1.5.7 and OpenBabel 2.4.1 software were used to perform molecular docking between the core targets and luteolin. The survival analysis and pan-cancer analysis of the core targets were performed in the GEPIA database.
ResultsA total of 449 targets of luteolin and 1 334 targets related to cervical cancer were obtained; there were 100 intersection targets between luteolin and cervical cancer, of which 24 were core targets, including MMP2, HRAS, MAPK1, AKT1, RHOA and PGR. GO and KEGG enrichment analyses revealed that the intersection targets participated in 455 biological processes, 70 cellular components, 119 molecular functions, and 143 KEGG signaling pathways. Molecular docking revealed a good binding of MMP2 with luteolin. The survival curves of cervical cancer patients showed that the risk ratios of RHOA, MAPK1, MMP2 and AKT1 genes were greater than 1, while those of HRAS and PGR were less than 1. Pan-cancer analysis showed that HRAS and MAPK1 were highly expressed in cervical cancer, and HRAS had significant expression differences.
ConclusionLuteolin treats cervical cancer through a multi-target and multi-pathway mechanism.
-
Keywords:
- network pharmacology /
- cervical cancer /
- luteolin /
- molecular mechanism /
- molecular docking /
- signaling pathway
-
年龄相关性白内障(ARC)为世界首位致盲性眼病,是一种衰老相关的慢性病,其发病率随年龄增长而增加[1-2]。目前,对ARC发生发展的具体分子机制仍然缺乏较全面的了解。长链非编码RNA(LncRNA)是长度大于200个核苷酸的非编码RNA, 可在基因的转录、转录后和翻译后等水平调控其表达,在多种生理学和病理学过程中发挥重要作用[3]。研究[4]发现大量LncRNA参与细胞衰老及相关信号通路的调控,影响关键细胞周期过程,如细胞增殖、分化、静止、衰老等。这类衰老相关LncRNA通过表观遗传调控、细胞增殖、端粒稳定性、蛋白质内稳态、细胞间通讯、干细胞池等调控衰老[5]。本研究探讨衰老相关LncRNA PINT对ARC发生发展的影响,现报告如下。
1. 材料与方法
1.1 研究对象
病例组(ARC组):在扬州大学附属医院眼科被诊断为ARC、接受白内障小切口手法碎核白内障手术患者。收集中度皮质性ARC、重度皮质性ARC、中度核性ARC、重度核性ARC各10眼。纳入标准: 患者年龄50~79岁; 最佳矫正视力低于0.5; 排除标准: 先天性、糖尿病性、外伤性、并发性、药物及中毒性白内障等患者。所有参加研究的患者均无高血压病、糖尿病、自身免疫性疾病及其他氧化损伤性相关疾病。
对照组: ①在扬州大学附属医院眼科被诊断为玻璃体视网膜疾病(如特发性黄斑前膜、特发性黄斑裂孔等),因年龄和手术等因素需要在玻璃体切除手术中同时行透明晶状体摘除者,共10眼。②扬州市红十字眼库捐献的新鲜眼球。纳入标准[6]: 年龄50~79岁; 晶状体透明。排除标准: 高血压病、糖尿病、自身免疫病及其他氧化损伤性相关疾病者。研究对象签署知情同意书。③人晶状体上皮细胞株SRA01/04(Thermo Fisher Scientific公司)。上述研究方案严格遵循赫尔辛基宣言,且经扬州大学医学院伦理委员会批准。
1.2 RNA提取、逆转录和实时荧光定量PCR
使用TRIzol试剂(Invitrogen, Carlsbad, CA, United States)从ARC和对照组细胞中提取总RNA, 然后使用PrimeScript RT Master Mix(Takara, 中国大连)、Power SYBR Green PCR Master Mix(Applied Biosystems, Irvine, CA, United States)在ABI 7500实时PCR系统上进行实时PCR分析。选用actin beta作为内参,每个实验重复3次。
1.3 细胞培养和处理
人晶状体上皮细胞株SRA01/04起源于人晶状体上皮细胞,将其培养在DMEM中,含有10%胎牛血清(FBS), 青霉素(100单位/mL)和链霉素(100 μg/mL)。所有细胞在37 ℃, 95%空气和5%二氧化碳的加湿培养箱中培养。氧化损伤组:当生长至80%~90%时,将细胞暴露于过氧化氢(H2O2)(Sigma-Aldrich Corp., St Louis, MO, USA)中,进行氧化损伤。将细胞在含2%FBS的DMEM中孵育过夜,然后在无血清DMEM中孵育30 min。用H2O2(100和200μmol/L)处理24 h后,收集细胞进行不同的测定。衰老组:当细胞生长至80%~90%时,将细胞暴露于小剂量H2O2进行衰老处理。将细胞在含2% FBS的DMEM中孵育过夜,然后在无血清DMEM中孵育30 min, 并用50 μmol/L H2O2培养至7、14、21 d。实验结束时,收取所有细胞并用于不同的检测。平行培养的对照细胞在类似的培养基中生长,不进行处理,作为所有实验的对照。
1.4 质粒构建、慢病毒包装和转染
使用pCDNA3.1载体(Genechem Technology Co., Shanghai, China)。过表达LINC-PINT: 通过PCR生成LINC-PINT序列并克隆到pcDNA3.1载体中。慢病毒包装:将pcDNA、pcDNA-LINC-PINT、si-NC、si-LINC-PINT分别转染进人晶状体上皮细胞株SRA01/04中,转染6 h后,更换新鲜培养液继续培养48 h, 检测转染效率。转染成功后,用于后续实验。
1.5 细胞增殖能力测定
利用CCK8检测试剂盒(Dojindo Laboratories, Kumamoto, Japan)检测各组细胞的生长增殖情况,分别向处理或未处理的细胞中加入10 μL CCK8试剂, 37 ℃避光孵育3 h, 酶标仪检测450 nm处的吸光度(OD值),实验重复3次。
1.6 抗氧化能力检测
总抗氧化能力(T-AOC)检测试剂盒(FRAP法)测定总抗氧化水平。在酸性环境下,还原铁-三吡啶三吖嗪(Fe3+-TPTZ)产生亚铁离子(Fe2+)形式的能力反映了总抗氧化能力。收集100~200万个细胞,加入1.0 mL预冷的提取液,超声充分破碎细胞并释放其中的抗氧化物, 4 ℃、10 000次/min, 离心5 min, 取上清液。样品的抗氧化能力以达到同样吸光度变化值所需的标准液离子浓度表示。
1.7 染色质免疫共沉淀(ChIP)技术
ChIP测定: 使用EZ-Magna ChIP A/G试剂盒(Millipore), 应用抗EZH2(Active Motif)和抗H3K27me3(Active Motif)。将SRA01/04细胞与1%甲醛交联8 min, 然后匀浆。使用Branson Digital Sonifier(Model 450; Branson Ultrasonics Corporation, CT, USA)的微尖探针将匀浆在2级超声处理4次,每次15 s, 每次脉冲之间在冰上间隔40 s, 产生长度为200~800碱基对(bp)的DNA片段。ChIP分析:将等量处理过的染色质添加到含有针对目标蛋白的固定化抗体或阴性对照正常兔IgG抗体的微孔中。在65 ℃温育90 min以逆转交联并洗脱DNA后,使用快速旋转柱进行DNA纯化,然后进行实时PCR检测。
1.8 统计分析
采用IBM SPSS Statistics 20.0软件进行数据分析,所有结果数据以平均值±标准差表示。2组之间的差异采用非配对的Student′s t检验或方差分析进行比较。P < 0.05为差异具有统计学意义。
2. 结果
2.1 LINC-PINT在ARC中的表达
使用高通量RNA序列分析、确定ARC组和对照组晶状体上皮细胞之间的差异表达。ARC组LINC-PINT的表达水平高于对照组,差异有统计学意义(P < 0.05)。LINC-PINT的表达水平与ARC分级呈正相关。见图 1。
2.2 衰老相关LncRNA与氧化损伤/衰老的关系
在晶状体上皮细胞中,氧化损伤组和衰老组中LINC-PINT表达水平高于对照组,差异有统计学意义(P < 0.05), 且分别呈现为剂量依赖性或时间依赖性的正相关,见图 2、图 3。
2.3 建立过表达/敲低模型
将pcDNA、pcDNA-LINC-PINT、si-NC、si-LINC-PINT分别转染进人晶状体上皮细胞SRA01/04中,通过实时荧光定量PCR检测转染前后细胞中相关基因的表达量变化,发现转入pcDNA-LINC-PINT后,靶基因CCNA2、CDK1、PCNA表达减少; 转入si-LINC-PINT后,靶基因CCNA2、CDK1、PCNA表达增加,见图 4。同时, LINC-PINT改变抗氧化损伤能力,见图 5。CCK8显示在LINC-PINT过表达的细胞中细胞增殖被抑制,见图 6。
2.4 衰老相关LncRNA与转录因子的调控机制
在模拟物和LINC-PINT过表达的细胞中进行ChIP, 测定表观遗传修饰物与LINC-PINT靶基因的启动子区域之间的相互作用。LINC-PINT与EZH2相互作用。LINC-PINT将EZH2招募到CDK1、CCNA2和PCNA基因的启动子上,导致H3K27三甲基化和靶基因的表观遗传学沉默。见图 7。
3. 讨论
LncRNA PINT是由p53诱导的核LncRNA, 直接与PRC2相互作用,且是PRC2特异靶基因中的组蛋白H3K27甲基化所必需的。研究[7]表明, LINC-PINT的功能依赖于PRC2的表达水平,并通过与PRC2相互作用调控转化生长因子-β(TGF-β)、丝裂原活化蛋白激酶(MAPK)和p53细胞通路中的蛋白质表达水平,而这些通路与氧化损伤、衰老和衰老相关性疾病有关。p53及其下游p21蛋白是重要的细胞周期相关蛋白,广泛参与了细胞的增殖、分化、凋亡等多种生理及病理过程。研究[4, 8]表明, LncRNA是p53/p21信号通路的重要组成部分,而调控表观遗传学变异的LncRNA PINT在p53/p21信号通路中扮演着重要的角色。p53能通过诱导LncRNA PINT的表达,与PRC2相互作用,从而终止基因转录,调控p53信号通路中蛋白质的表达水平,调控细胞周期进程,从而影响机体的衰老以及衰老相关疾病的发生发展。
ARC与表观遗传因素密不可分。研究[9-10]报道, LncRNA与许多眼病相关,如青光眼、后发性白内障、糖尿病性视网膜病变、增生性玻璃体视网膜病变、眼部肿瘤等。其中有报道[11]表明LncRNA PAUPAR通过调控组蛋白H3K4的去甲基化进而抑制葡萄膜黑色素瘤的发生。作者的前期研究[12-13]提示,在人晶状体上皮细胞株SRA01/04中,高浓度短时间的氧化损伤可诱导氧化损伤模型,而低浓度长时间的氧化损伤可诱导衰老模型。ARC的衰老发病机制与氧化损伤机制紧密相扣,氧化损伤相关的表观遗传学改变与ARC密切相关[14]。因此,衰老相关LncRNA可能通过参与表观遗传学调控影响细胞衰老,进而引起ARC。
本研究初步阐明了LncRNA PINT可能通过p53/p21通路调控相关基因的表观遗传改变,参与ARC的发生发展,揭示了衰老相关LncRNA在ARC发病机制中起到重要作用,可作为ARC防治的新靶点。本研究从表观遗传学水平探讨ARC发病机制,为ARC防治提供新思路,有助于研发抗ARC药物,为进一步深入了解白内障的发病机理提供了理论基础和方向。
-
-
[1] SUNG H, FERLAY J, SIEGEL R L, et al. Global cancer statistics 2020: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA Cancer J Clin, 2021, 71(3): 209-249. doi: 10.3322/caac.21660
[2] SINGH D, VIGNAT J, LORENZONI V, et al. Global estimates of incidence and mortality of cervical cancer in 2020: a baseline analysis of the WHO Global Cervical Cancer Elimination Initiative[J]. Lancet Glob Health, 2023, 11(2): e197-e206.
[3] REVATHIDEVI S, MURUGAN A K, NAKAOKA H, et al. APOBEC: a molecular driver in cervical cancer pathogenesis[J]. Cancer Lett, 2021, 496: 104-116.
[4] RAHANGDALE L, MUNGO C, O'CONNOR S, et al. Human papillomavirus vaccination and cervical cancer risk[J]. BMJ, 2022, 379: e070115.
[5] GOPU P, ANTONY F, CYRIAC S, et al. Updates on systemic therapy for cervical cancer[J]. Indian J Med Res, 2021, 154(2): 293-302.
[6] 程志新, 公瑞, 张玥, 等. 基于数据挖掘的臁疮古医案内治法用药规律研究[J]. 实用临床医药杂志, 2023, 27(10): 25-30. doi: 10.7619/jcmp.20230525 [7] FRANZA L, CARUSI V, NUCERA E, et al. Luteolin, inflammation and cancer: Special emphasis on gut microbiota[J]. Biofactors, 2021, 47(2): 181-189.
[8] NABAVI S F, BRAIDY N, GORTZI O, et al. Luteolin as an anti-inflammatory and neuroprotective agent: a brief review[J]. Brain Res Bull, 2015, 119(Pt A): 1-11.
[9] RAKOCZY K, KACZOR J, SOŁTYK A, et al. Application of luteolin in neoplasms and nonneoplastic diseases[J]. Int J Mol Sci, 2023, 24(21): 15995.
[10] CHEN Y H, WU J X, YANG S F, et al. Synergistic combination of luteolin and Asiatic acid on cervical cancer in vitro and in vivo[J]. Cancers, 2023, 15(2): 548.
[11] DING L L, CHEN H, BI G L, et al. Improved anti-cancer effects of luteolin@ZIF-8 in cervical and prostate cancer cell lines[J]. Heliyon, 2024, 10(6): e28232.
[12] LI X, WEI S Z, NIU S Q, et al. Network pharmacology prediction and molecular docking-based strategy to explore the potential mechanism of Huanglian Jiedu Decoction against sepsis[J]. Comput Biol Med, 2022, 144: 105389.
[13] LIU X F, OUYANG S S, YU B, et al. PharmMapper server: a web server for potential drug target identification using pharmacophore mapping approach[J]. Nucleic Acids Res, 2010, 38(Web Server issue): W609-W614.
[14] GALLO K, GOEDE A, PREISSNER R, et al. SuperPred 3. 0: drug classification and target prediction-a machine learning approach[J]. Nucleic Acids Res, 2022, 50(W1): W726-W731.
[15] DAINA A, MICHIELIN O, ZOETE V. SwissTargetPrediction: updated data and new features for efficient prediction of protein targets of small molecules[J]. Nucleic Acids Res, 2019, 47(W1): W357-W364.
[16] DAINA A, ZOETE V. Application of the SwissDrugDesign online resources in virtual screening[J]. Int J Mol Sci, 2019, 20(18): 4612.
[17] STELZER G, ROSEN N, PLASCHKES I, et al. The GeneCards Suite: From Gene Data Mining to Disease Genome Sequence Analyses[J]. Curr Protoc Bioinformatics, 2016, 54: 30-33.
[18] AMBERGER J S, BOCCHINI C A, SCOTT A F, et al. OMIM. org: leveraging knowledge across phenotype-gene relationships[J]. Nucleic Acids Res, 2019, 47(D1): D1038-D1043.
[19] STEVENS T, SANGKUHL K, BROWN J T, et al. PharmGKB summary: methylphenidate pathway, pharmacokinetics/pharmacodynamics[J]. Pharmacogenet Genomics, 2019, 29(6): 136-154.
[20] DENNIS G, SHERMAN B T, HOSACK D A, et al. DAVID: database for annotation, visualization, and integrated discovery[J]. Genome Biol, 2003, 4(9): R60.
[21] MUEGGE I, HU Y. Recent advances in alchemical binding free energy calculations for drug discovery[J]. ACS Med Chem Lett, 2023, 14(3): 244-250.
[22] WANG X, WANG Z Y, ZHENG J H, et al. TCM network pharmacology: a new trend towards combining computational, experimental and clinical approaches[J]. Chin J Nat Med, 2021, 19(1): 1-11.
[23] NOGALES C, MAMDOUH Z M, LIST M, et al. Network pharmacology: curing causal mechanisms instead of treating symptoms[J]. Trends Pharmacol Sci, 2022, 43(2): 136-150. doi: 10.1016/j.tips.2021.11.004
[24] HAN X, WANG C, LI R, et al. Network target theory and network pharmacology: Next generation drug research in medicine using AI models[J]. Int J Clin Pharmacol Ther, 2024, 62(4): 151-154.
[25] 许继升, 宋爱莉, 王静, 等. 基于数据挖掘和网络药理学探讨宋爱莉教授治疗肉芽肿性小叶性乳腺炎的经验[J]. 实用临床医药杂志, 2023, 9(5): 49-54. doi: 10.7619/jcmp.20222860 [26] 金雪宁, 林飞, 冯心怡, 等. 基于网络药理学的多西他赛治疗绒毛膜癌的作用机制研究[J]. 实用临床医药杂志, 2022, 8(1): 18-21. doi: 10.7619/jcmp.20213494 [27] ZHANG Q, YANG M Y, QU Z, et al. Corrigendum to "Autophagy prevention sensitizes AKTi-1/2-induced anti-hepatocellular carcinoma cell activity in vitro and in vivo" [Biochem. Biophys. Res. Commun. (2016) 480(3) 334-340][J]. Biochem Biophys Res Commun, 2021, 565: 99.
[28] YANG Y F, HUANG L, ZHANG N, et al. SUMOylation of annexin A6 retards cell migration and tumor growth by suppressing RHOU/AKT1-involved EMT in hepatocellular carcinoma[J]. Cell Commun Signal, 2024, 22(1): 206.
[29] 金雪宁, 林飞, 冯心怡, 等. 基于网络药理学的多西他赛治疗绒毛膜癌的作用机制研究[J]. 实用临床医药杂志, 2022, 26(1): 18-21. doi: 10.7619/jcmp.20213494 [30] 罗字呈, 杨桃, 陈江, 等. 基于网络药理学及生物信息学探讨藤梨汤治疗结直肠癌的作用机制研究[J]. 空军军医大学学报, 2024(3): 311-318. [31] 曾炼, 杨桃, 陈江, 等. 基于网络药理学和分子对接探讨艾迪注射液治疗结直肠癌作用机制研究[J]. 空军军医大学学报, 2024, 45(03): 319-325. [32] 甘丽华, 李志敏, 何泉, 杨坤, 郭超峰. 基于分子对接技术与网络药理学分析方法的半夏-黄连药对治疗胃食管反流病的作用机制研究[J]. 实用临床医药杂志, 2021, 25(21): 1-6. doi: 10.7619/jcmp.20211655 [33] LI L, SHEN F R, CHENG Q X, et al. SLC5A3 is important for cervical cancer cell growth[J]. Int J Biol Sci, 2023, 19(9): 2787-2802.
[34] LI S, MA Y M, ZHENG P S, et al. GDF15 promotes the proliferation of cervical cancer cells by phosphorylating AKT1 and Erk1/2 through the receptor ErbB2[J]. J Exp Clin Cancer Res, 2018, 37(1): 80.
[35] LIU Z, LIU F, WANG F, et al. CircZNF609 promotes cell proliferation, migration, invasion, and glycolysis in nasopharyngeal carcinoma through regulating HRAS via miR-338-3p[J]. Mol Cell Biochem, 2021, 476(1): 175-186.
[36] PAN Y B, GU Y M, LIU T H, et al. Epitranscriptic regulation of HRAS by N6-methyladenosine drives tumor progression[J]. Proc Natl Acad Sci U S A, 2023, 120(14): e2302291120.
[37] RIEKE D T, SCHR?DER S, SCHAFHAUSEN P, et al. Targeted treatment in a case series of AR+, HRAS/PIK3CA co-mutated salivary duct carcinoma[J]. Front Oncol, 2023, 13: 1107134.
[38] BAI H, SONG M, JIAO R, et al. DUSP7 inhibits cervical cancer progression by inactivating the RAS pathway[J]. J Cell Mol Med, 2021, 25(19): 9306-9318.





 下载:
下载:







 苏公网安备 32100302010246号
苏公网安备 32100302010246号
