Associations of transforming growth factor-β and meteorin-like protein with macroangiopathy in type 2 diabetes mellitus
-
摘要:目的
探讨转化生长因子-β(TGF-β)和镍纹样蛋白(Metrnl)与2型糖尿病(T2DM)大血管病变的相关性。
方法选取T2DM患者106例, 按颈动脉内中层厚度(CIMT)分为单纯T2DM组50例(CIMT≤1.0 mm)、T2DM合并大血管病变组56例(CIMT>1.0 mm), 并随机选取同期体检的健康受试者52例为健康对照组。比较3组外周血清中的TGF-β、Metrnl水平,分析各代谢指标与T2DM大血管病变的相关性。
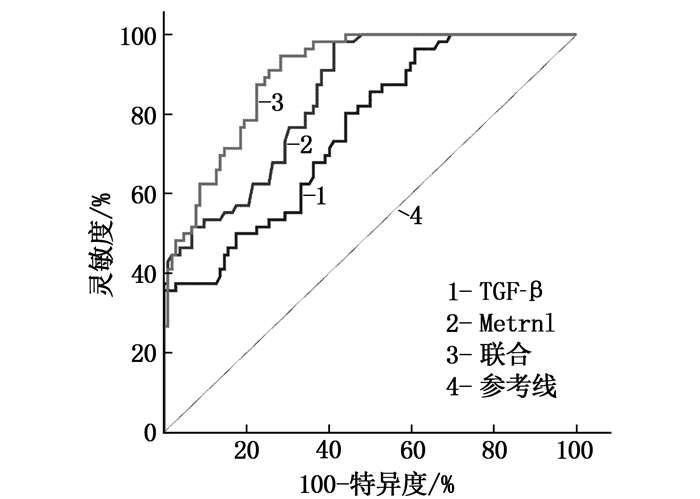
结果T2DM合并大血管病变组TGF-β、Metrnl水平低于单纯T2DM组和健康对照组,差异有统计学意义(P < 0.05); 血清TGF-β与Metrnl水平呈正相关(P < 0.05); 收缩压、吸烟、低密度脂蛋白胆固醇(LDL-C)、HOMA-IR是T2DM大血管病变的危险因素, Mernl、TGF-β是T2DM大血管病变的保护因素(P < 0.05); 受试者工作特征(ROC)曲线分析结果显示, TGF-β和Metrnl预测的曲线下面积(AUC)分别为0.757、0.846, TGF-β联合Metrnl预测的AUC为0.901。
结论血清TGF-β和Metrnl水平降低与T2DM大血管病变的发生相关, TGF-β联合Metrnl检测对T2DM大血管病变的早期评估具有重要意义。
Abstract:ObjectiveTo investigate the correlations of transforming growth factor-β (TGF-β)and meteorin-like protein(Metrnl) with macroangiopathy in type 2 diabetes mellitus.
MethodsA total of 106 patients with T2DM were divided into simple T2DM group (n=50, CIMT ≤ 1.0 mm), T2DM combined with macroangiopathy disease group (n=56, CIMT>1.0 mm), and 52 healthy subjects who underwent physical examination at the same time were randomly selected as healthy control group. Metrnl and TGF-β levels in peripheral serum in three groups were compared. The correlations of metabolic indexes with T2DM macroangiopathy were compared between the three groups.
ResultsThe levels of TGF-β and Metrnl in the T2DM combined with macroangiopathy disease group were significantly lower than those in the T2DM simple group and healthy control group (P < 0.05); there was a positive correlation between TGF-β and Metrnl level (P < 0.05); systolic blood pressure, smoking, low-density lipoprotein cholesterol (LDL-C) and HOMA-IR were risk factors for T2DM macroangiopaopathy, while Metrnl and TGF-β were protective factors for T2DM macroangiopaopathy (P < 0.05); receiver operating characteristic (ROC) curve analysis showed that the area under the curve (AUC) predicted by TGF-β and Metrnl were 0.757 and 0.846, respectively, the AUC predicted by TGF-β combined with Metrnl was 0.901.
ConclusionSerum TGF-β and decreased Metrnl levels are associated with the occurrence of T2DM macrovascular disease. Detection of TGF-β combined with Metrnl is of great significance for the early evaluation of T2DM macrovascular disease.
-
在人口老龄化和代谢危险因素的双重压力下,中国居民的心血管疾病风险持续增加,心肌梗死患者的病死率亦逐渐上升[1]。心肌梗死是指心肌缺血性坏死,通常是在冠状动脉病变的基础上发生冠状动脉血供减少或中断,心肌细胞因严重且持续的缺血缺氧而发生坏死[2]。心肌细胞的死亡会引发一系列免疫炎症反应,这些免疫炎症反应介导的受损心肌修复在梗死后心室重构、心功能和患者结局等方面起着关键作用[3]。急性心肌梗死的诊断主要依据临床表现、血清生物学标志物和心电图,其中临床常用的生物学标志物包括肌红蛋白、肌酸激酶-MB、心肌肌钙蛋白I和心肌肌钙蛋白T[4-5]。然而,这些标志物水平在心力衰竭、肾衰竭和甲状腺疾病患者中也同样升高,故结合多组学平台探寻更多特异性的生物学标志物非常必要[6-7]。外周血单个核细胞(PBMCs)包括T细胞、B细胞、单核细胞、树突状细胞和自然杀伤细胞,在免疫应答、免疫监测和免疫治疗中发挥着重要作用[8]。本研究基于PBMCs分析心肌梗死和梗死恢复过程中潜在的关键基因和生物学改变,旨在为心肌梗死的诊断、评估、管理和预后分析提供新的见解。
1. 材料和方法
1.1 数据集获取
从GEO数据库中下载关于心肌梗死PBMCs的转录组测序芯片数据集GSE59867[9-10], 芯片注释平台为GPL6244, 芯片信息为Affymetrix Human Gene 1.0 ST Array。该数据集收集了111例ST段抬高型心肌梗死(STEMI)患者入院、出院、心肌梗死后1个月、心肌梗死后6个月时的外周血样本数据。对照组为46例无心肌梗死病史的稳定型冠心病患者。基于芯片注释平台对数据集进行基因名转换,用于后续共表达网络的构建。
1.2 加权基因共表达网络分析(WGCNA)
通过WGCNA R包筛选与心肌梗死有关的易感基因模块[11]。WGCNA是一种常用的生物信息学方法,可以识别具有相似表达模式的基因模块,分析基因模块与样本表型之间的关系,绘制基因模块中的调控网络,识别关键模块和调控基因[12-13]。首先,选取数据集中方差变异程度最高的前3 000个基因构建共表达网络; 其次,利用R函数pickSoftThreshold计算软阈值1~20; 然后,对这些基因进行层次聚类和动态树切割来划分模块,并合并相似的模块(阈值为0.25); 最后,计算模块与疾病之间的Spearman相关系数,进而确定易感基因模块。为了探讨所选模块的潜在生物学功能,通过ClusterProfiler R包对模块中的基因进行基因本体论(GO)分析和京都基因与基因组百科全书(KEGG)通路分析[14]。GO分析包括生物过程(BP)、分子功能(MF)和细胞成分(CC)。通过Ggplot2 R包进行结果展示。
1.3 基因差异分析和转录因子预测
使用Limma R包进行基因差异分析[15]。差异表达基因(DEGs)的筛选标准为矫正后P < 0.05, |log2(倍数变化)|>0.5。基于ClusterProfiler R包对DEGs进行GO分析和KEGG通路分析, P < 0.05表示显著富集。对上述易感基因模块和DEGs进行交集分析,通过ChEA3数据库对交集基因进行转录因子预测。ChEA3是一种转录因子富集分析工具,整合了多个数据库,如ENCODE、GTEx、ARCHS4等数据库[16]。由于ChEA3数据库并未对预测结果可靠度进行严格划分,参考相关研究[16-17]结论,本研究对平均排名分≤30分的共同转录因子进行结果展示。基于STRING数据库构建交集基因的蛋白相互作用网络,并将交互评分>0.4分的数据导入Cytoscape 3.8.2软件进行可视化处理[18-19]。
1.4 关键基因的确定和受试者工作特征(ROC) 曲线分析
LASSO回归分析是常用的筛选变量的压缩估计方法,通过构造惩罚函数获得较为精炼的模型,使得其压缩一些回归系数,同时设定一些回归系数为0,因此保留了子集收缩的优点,是一种处理具有复共线性数据的有偏估计,常被用于协变量筛选[20]。基于glmnet R包对交集基因中存在蛋白相互作用的基因进行LASSO回归分析,进一步筛选关键基因[21]。采用10倍交叉验证法选择惩罚项(λ), 并选择最小二项偏差在1个标准误差内的最简单模型的λ值(lambda.1se)。通过pROC R包对关键基因进行ROC曲线分析,计算曲线下面积(AUC), 评估关键基因对心肌梗死的诊断价值[22]。
1.5 基因集富集分析(GSEA)和外部数据集验证
根据每个关键基因的表达量将数据集划分为高表达组和低表达组,通过GSEA探讨高表达组和低表达组之间潜在的生物学功能变化,将特征基因集作为预先定义的基因集。作为一种常见的生物信息学方法, GSEA可评估一个预先定义的基因集的基因在与表型相关度排序的基因表中的分布趋势,从而判断其对表型的影响[23]。从GEO数据库中选取另一个数据集GSE123342, 将其用于验证关键基因的表达。GSE123342数据集包括急性心肌梗死(65例)、梗死后30 d(64例)、梗死后1年(37例)、稳定型冠心病(22例)样本数据以及4例技术重复样本数据。
2. 结果
2.1 易感基因模块的确定
根据无标度网络原理,选取10作为共表达网络的软阈值,见图 1A。通过层次聚类和动态树切割,将前3 000个基因划分为15个基因模块,见图 1B。剔除聚类失败的灰色模块,棕色模块的总体表达与心肌梗死的相关性最高(r=0.4), 且与出院至心肌梗死后6个月的演变过程呈负相关(r=-0.33), 见图 1C。棕色模块中的共表达基因伴随着心肌梗死的发生呈现高表达趋势,而在心肌梗死恢复过程中则呈现低表达趋势。由此提示,棕色模块可能是易感基因模块,其中包含243个基因,即ABHD5、ACER3、ACP3、ACSL1、ADAM17、ADAM9、ADM、AHR、ALCAM、ALDH2、ALPK1、ANKRD50、ANO5、AQP9、ARHGAP24、ARHGAP29、ARRDC4、ASPH、ATP6V0A1、ATP6V1A、B3GNT5、BACH1、BLVRB、BST1、C3AR1、C5、C9orf72、CALCRL、CAPG、CAPZA2、CARD6、CASP1、CASP5、CCDC88A、CCR2、CD14、CD163、CD1D、CD33、CD36、CD63、CLEC1A、CLEC4A、CLEC4D、CLEC4E、CLEC7A、CLMN、CNTLN、CPED1、CPM、CPNE8、CPVL、CR1、CR1L、CREG1、CRISPLD2、CSTA、CTNNA1、CTSH、CYBRD1、CYP1B1、CYP1B1-AS1、DACH1、DDIAS、DOCK4、DOCK5、DRAM1、DSC2、DSE、DUSP6、EDNRB、ENTPD1、ERLIN1、EVI5、F5、FAM114A1、FAM13A、FAM151B、FAM20A、FAM198B、FAR2、FBN2、FBP1、FCGR2A、FGD4、FGD6、FLT3、FLVCR2、FMO5、FPR1、FPR2、FRRS1、FUCA1、FUCA2、GAPT、GAS2L3、GCA、GIMAP8、GLT1D1、GPAT3、GPR141、HAL、HGF、HMGB2、HNMT、HORMAD1、HP、HPSE、IDH1、IFNGR1、IL13RA1、IL15、IL18、IMPA2、IRAK3、JAK2、KCNJ15、KCNJ2、KIF13A、KLHL8、KYNU、LHFPL2、LILRA5、LIN7A、LIPN、LMNB1、LPCAT2、LRMDA、LRRK2、LTA4H、LY86、LYVE1、MAP3K20、MCEMP1、ME1、METTL7A、METTL7B、MFSD1、MGST1、MGST2、MILR1、MNDA、 MOSPD2、MS4A4A、MSRB1、MTARC1、MYOF、NAAA、NAIP、NETO2、NLN、NLRC4、NPL、OLFML2B、P2RX7、P2RY13、PGD、PIP4P2、PLA2G4A、PLBD1、PLD1、PLIN2、PLSCR1、PPARG、PPT1、PRRG4、PSTPIP2、PYGL、QPCT、RAB39A、RALB、RBP7、RGL1、RNASE2、RNASE6、RNF141、RNF217、RRAGD、S100A12、S100A9、S100Z、SAT2、SEMA3C、SEPTIN10、SERPINB1、SERPINB2、SESTD1、SGMS2、SH3PXD2B、SHTN1、SIPA1L2、SIRPD、SLC15A2、SLC1A3、SLC22A15、SLC22A4、SLC26A8、SLC31A1、SLC36A4、SLC7A7、SLC8A1、SLITRK4、SMAD1、SMPDL3A、SNX10、SOD2、SORT1、SPATA6、ST3GAL6、ST6GALNAC3、STEAP4、SULT1B1、TASL、TBC1D12、TCN2、TDRD9、TFEC、TGFBI、TLR1、TLR2、TLR4、TLR5、TLR6、TLR7、TLR8、TM6SF1、TMEM144、TMEM167A、TMTC2、TNFAIP6、TNFSF13B、TPST1、TSPO、UBE2D1、UGGT2、VNN2、VNN3、WASF1、WDFY3、WLS、ZC3H12C、ZFYVE16、ZNF438。
棕色模块GO分析最显著的结果分别是骨髓白细胞激活(BP)、囊泡(CC)和模式识别受体活性(MF), 见图 1D; KEGG分析结果则主要为感染、免疫炎症等相关生物学过程的改变,包括Toll样受体信号通路等,见图 1E。
2.2 基因差异分析
进一步对数据集进行基因差异分析,根据筛选标准共得到142个DEGs(图 2A), 其中上调的DEGs有77个,下调的DEGs有65个,见表 1。BP分析结果主要包括免疫反应、免疫系统过程和免疫效应过程, CC分析结果包括分泌颗粒、分泌小泡和细胞质小泡部分等, MF分析结果主要包括信号受体活性、分子传感器活性和碳水化合物结合,见图 2B。KEGG分析提示,这些DEGs主要涉及免疫、炎症和感染的相关生物学改变,见图 2C。DEGs与上述棕色模块存在35个交集基因,见图 2D。基于ChEA3数据库对35个交集基因进行共同转录因子预测,其中平均排名分≤30分的共同转录因子分别是CREB5、MTF1、NFE4、SPI1、ZNF467、NFE2、TFEC、MXD1、NR1H3、BORCS8MEF2B、NFIL3和CEBPE, 见表 2。构建蛋白互作网络,其中有18个基因存在蛋白质相互作用,见图 2E。
表 1 差异基因与交集基因上调DEGs 下调DEGs 与棕色模块交集的DEGs ACSL1、ADM、AQP9、ASGR2、BLVRB、BST1、CCR1、CCR2、CD14、CD163、CES1、CES1P1、CR1、CYP1B1、CYP27A1、DSC2、DYSF、ECRP、EGR1、EGR2、FAM20A、FAM20C、FLVCR2、FMN1、FOLR3、FPR1、FPR2、FAM198B、GPR162、HLX、HP、IFITM3、IL1R2、KCNJ15、LILRA3、LILRA5、LILRB4、LRP1、MAFB、MCEMP1、MERTK、MIR21、MS4A4A、MTARC1、NFE2、NRG1、PADI2、PLB1、PLBD1、PPARG、QPCT、RNASE1、RNASE2、S100A12、S100A9、SASH1、SERPINB10、SERPINB2、SH3PXD2B、SIGLEC16、SIGLEC9、SLED1、SOCS3、ST14、STAB 1、STEAP4、TBC1D2、TCN2、TDRD9、TMEM150B、TMEM176A、TMEM176B、TNFAIP6、TREM1、TRIB1、TSPO、VSIG4 ARAP2、BIRC3、C12orf75、CD160、CD3G、CENPK、CEP78、CLC、CLEC2D、CPA3、DTHD1、EPB41L4A、FAM169A、FCRL6、FGFBP2、GBP4、GNLY、HRH4、IL18RAP、JAKMIP2、KLRA1P、KLRC1、KLRC3、KLRC4-KLRK1、KLRD1、KLRF1、KLRG1、KPNA5、MIR29A、MS4A3、MYBL1、MYO6、ODF2L、PDGFD、PRSS23、RNU5A-1、RORA、SAMD3、SCARNA9、SH2D1A、SLC4A4、SLCO4C1、SNORA16B、SNORA20、SNORA75、SNORD20、SNORD28、SNORD30、SNORD45C、SNORD50A、SNORD54、SNORD58A、SNORD59A、SNORD59B、SNORD61、SNORD81、SNORD82、SYTL2、TARP、TBC1D19、TFDP2、TGFBR3、VPS13A、ZNF675、ZNF737 ACSL1、ADM、AQP9、BLVRB、BST1、CCR2、CD14、CD163、CR1、CYP1B1、DSC2、FAM20A、FLVCR2、FPR1、FPR2、FAM198B、HP、KCNJ15、LILRA5、MCEMP1、MS4A4A、MTARC1、PLBD1、PPARG、QPCT、RNASE2、S100A12、S100A9、SERPINB2、SH3PXD2B、STEAP4、TCN2、TDRD9、TNFAIP6、TSPO 表 2 共同转录因子预测结果(平均排名分≤30分)排名 转录因子 平均得分/分 转录因子数据库计算方法与得分 重叠基因 1 CREB5 7.667 ARCHS4 Coexpression, 5分; Enrichr Queries, 14分; GTEx Coexpression, 4分 CR1、STEAP4、TNFAIP6、ACSL1、AQP9、FPR1、MCEMP1、ADM、FPR2、LILRA5、BST1、QPCT、PLBD1、CYP1B1、CD14、S100A9、CCR2 2 MTF1 8.500 ARCHS4 Coexpression, 6分; GTEx Coexpression, 11分 CR1、SERPINB2、TNFAIP6、ACSL1、AQP9、FPR1、FPR2、LILRA5、BST1、PLBD1、CYP1B1、FLVCR2、CD14、S100A9 3 NFE4 9.000 GTEx Coexpression, 9分 BST1、CR1、AQP9、FPR1、PLBD1、MCEMP1、FPR2、LILRA5、CCR2 4 SPI1 9.667 Literature ChIP-seq, 5分; ARCHS4 Coexpression, 11分; ENCODE ChIP-seq, 8分; Enrichr Queries, 11分; ReMap ChIP-seq, 5分; GTEx Coexpression, 18分 CD163、CR1、STEAP4、SERPINB2、ACSL1、AQP9、FPR1、TDRD9、MCEMP1、FPR2、RNASE2、LILRA5、MS4A4A、BST1、QPCT、PLBD1、CYP1B1、CD14、S100A9、CCR2 5 ZNF467 19.333 ARCHS4 Coexpression, 18分; Enrichr Queries, 30分; GTEx Coexpression, 10分 CR1、AQP9、FPR1、MCEMP1、ADM、FPR2、LILRA5、BST1、PLBD1、CYP1B1、BLVRB、CD14、S100A9、CCR2 6 NFE2 21.000 ARCHS4 Coexpression, 16分; ENCODE ChIP-seq, 78分; Enrichr Queries, 1分; ReMap ChIP-seq, 8分; GTEx Coexpression, 2分 CR1、SERPINB2、AQP9、FPR1、MCEMP1、ADM、FPR2、RNASE2、LILRA5、BST1、TCN2、QPCT、PLBD1、S100A12、BLVRB、TSPO、CYP1B1、PPARG、CD14、S100A9、CCR2 7 TFEC 21.667 ARCHS4 Coexpression, 21分; Enrichr Queries, 4分; GTEx Coexpression, 40分 CD163、CR1、SERPINB2、TNFAIP6、ACSL1、AQP9、FPR1、MS4A4A、BST1、TCN2、FLVCR2、PPARG、CD14、S100A9、CCR2 8 MXD1 23.000 ARCHS4 Coexpression, 4分; Enrichr Queries, 9分; GTEx Coexpression, 56分 CR1、SERPINB2、STEAP4、ACSL1、AQP9、FPR1、ADM、FPR2、LILRA5、BST1、PLBD1、CYP1B1、CD14、S100A9 9 NR1H3 26.600 Literature ChIP-seq, 10分; ARCHS4 Coexpression, 17分; Enrichr Queries, 10分; ReMap ChIP-seq, 36分; GTEx Coexpression, 60分 CD163、STEAP4、TCN2、ACSL1、FLVCR2、CYP1B1、PPARG、ADM、CD14、S100A9、MS4A4A 10 BORCS8MEF2B 29.000 ARCHS4 Coexpression, 29分 AQP9、FPR1、TSPO、FPR2、S100A9、LILRA5 11 NFIL3 30.000 ARCHS4 Coexpression, 1分; Enrichr Queries, 21分; GTEx Coexpression, 68分 CR1、TNFAIP6、ACSL1、AQP9、FPR1、ADM、FPR2、LILRA5、MS4A4A、BST1、CYP1B1、PLBD1、CD14、S100A9 12 CEBPE 30.000 ARCHS4 Coexpression, 31分; Enrichr Queries, 12分; GTEx Coexpression, 47分 BST1、FPR1、PLBD1、ADM、MCEMP1、FPR2、CD14、RNASE2、S100A9、CCR2、LILRA5 计算方法包括ARCHS4 Coexpression、ENCODE ChIP-seq、Enrichr Queries、GTEx Coexpression、Literature ChIP-seq、ReMap ChIP-seq; 平均得分为各计算方法得分之和除以计算方法数量所得之商。 2.3 关键基因的确定
对18个存在蛋白相互作用的基因进一步行LASSO回归分析,这些基因不同惩罚参数值所对应的系数见图 3A。本研究选取最小二项偏差在1个标准误差内的λ值(lambda.1se, 6个基因),该λ值提供更精简的模型,见图 3B。ROC曲线分析显示, CD163、RNASE2、HP、FAM20A、MCEMP1和FAM198B基因表达水平对心肌梗死的发生均具有良好的诊断价值, AUC分别为0.831、0.798、0.775、0763、0.866和0.829, 见图 3C。与对照组稳定型冠心病患者相比,这些关键基因的表达水平在入院时心肌梗死患者中显著上升,并在出院时、心肌梗死后1个月、心肌梗死后6个月逐步下降,见图 3D。
2.4 GSEA结果和验证
GSEA结果提示,这些关键基因涉及的生物学改变主要与糖脂代谢、活性氧、免疫炎症等有关,见图 4。这些基因在外部数据集中大多也存在差异表达。基于外部验证数据集GSE123342, 本研究同样发现,与稳定型冠心病相比, HP、FAM198B、CD163、FAM20A、MCEMP1表达水平在急性心肌梗死发生时显著上升,而RNASE2仅表现出上升趋势,见图 5。
3. 讨论
本研究基于PBMCs转录组学的变化探讨心肌梗死及梗死恢复过程中可能存在的生物学改变和潜在标志物, WGCNA和DEGs分析结果显示,心肌梗死过程伴随着免疫炎症紊乱。基于LASSO分析,本研究从上述2种分析方法得到的共同基因中鉴定出6个关键基因,即 CD163、RNASE2、HP、FAM20A、MCEMP1和FAM198B。ROC曲线分析结果提示,这些关键基因对心肌梗死的发生均具有较高的诊断价值。本研究还发现,在梗死恢复过程中,这些基因的表达呈现明显下降趋势。GSEA分析结果表明,这些关键基因涉及的生物学改变主要与糖脂代谢、活性氧、免疫炎症等有关。
CD163分子是Ⅰ型膜蛋白,蛋白表达限于单核细胞/巨噬细胞系, CD163抗原特异性释放机制可能在炎症调节过程中起重要作用。在动脉粥样硬化过程中, CD163+巨噬细胞能促进血管生成和增加血管通透性,并伴随炎症反应;此外,破裂的冠状动脉斑块中 CD163 的表达增加了心肌梗死和冠心病发生风险[24]。 RNASE2 编码的蛋白质是非分泌型核糖核酸酶,属于胰核糖核酸酶家族[25]。 FAM20A 是激酶编码基因家族的成员,本身不具有激酶活性,通过与家族成员 FAM20C 形成复合物,增强 FAM20C 的激酶活性,从而使分泌通路内的蛋白磷酸化[26]。 MCEMP1 又称 C19ORF59 , 可编码表达于肥大细胞、巨噬细胞等的跨膜蛋白[27]。研究[27]表明, MCEMP1 基因可能是卒中诊断和预后评估的新生物标志物。生物信息学研究[28-29]提示, RNASE2、FAM20A和 MCEMP1 可能是心肌梗死的关键基因,然而这些基因在心肌梗死及梗死恢复过程中的具体作用机制有待进一步研究。HP基因编码一种前蛋白,经处理后产生α链和β链,结合为四聚体产生触珠蛋白,其基因型与急性心肌梗死的发病风险密切相关,并且能决定心肌梗死面积[30-31]。 FAM198B(GASK1B) 是目前未知功能的新基因,可能编码定位在高尔基体上的膜结合糖蛋白,被认为参与癌症的转移与进展[32]。本研究分析结果显示, FAM198B 可能是心肌梗死和反映心肌梗死恢复的关键基因。
本研究尚存在一定局限性: 首先,本研究基于GEO数据库中下载的心肌梗死患者PBMCs的测序数据集进行分析,还需要进一步开展分子生物学实验来验证这些基因在心肌梗死中的表达及其可能的生物学机制; 其次,本研究仅分析了现有的样本数据,未来还需基于更大的样本规模和更详细的样本类型进一步深入研究; 最后,由于缺乏临床资料,本研究未分析这些关键基因的表达与心肌梗死患者年龄、性别、射血分数等临床指标的相关性。
综上所述,本研究基于PBMCs确定了6个可能与心肌梗死密切相关的关键基因,即 CD163、RNASE2、HP、FAM20A、MCEMP1和FAM198B, 为心肌梗死的诊断、评估、管理和预后分析提供了新的思路。
-
表 1 3组研究对象一般资料比较(x±s)[n(%)]
一般资料 单纯T2DM组(n=50) T2DM合并大血管病变组(n=56) 健康对照组(n=52) 年龄/岁 47.88±14.46 52.52±12.50 46.79±13.11 性别 男 34(68.00) 37(66.07) 33(63.46) 女 16(32.00) 19(33.93) 19(36.54) 收缩压/mmHg 125.42±7.17* 130.48±8.82*# 120.64±8.01 舒张压/mmHg 71.62±6.62 70.55±5.95 70.54±7.22 身高/cm 167.13±7.57 165.79±8.28 167.77±9.90 体质量/kg 68.16±11.62 70.67±12.51*# 64.43±12.60 体质量指数/(kg/m2) 24.28±3.05* 25.63±3.56*# 22.73±2.94 腰围/cm 89.9±9.07* 91.72±8.48*# 80.65±19.11 臀围/cm 96.76±6.81* 97.62±5.83*# 92.69±6.13 腰臀比 0.93±0.07 0.94±0.06 0.88±0.22 病程/年 4.94±2.19* 7.95±2.73*# — 吸烟 16(32.00) 28(50.00) 16(30.77) 与健康对照组比较, *P < 0.05; 与单纯T2DM组比较, #P < 0.05。 表 2 3组生化指标比较[M(P25, P75)](x±s)
指标 单纯T2DM组(n=50) T2DM合并大血管病变组(n=56) 健康对照组(n=52) FBG/(mmol/L) 7.48(6.23, 9.33)* 7.17(6.35, 10.02)*# 4.66(4.43, 4.99) CRP/(mg/dL) 1.66(0.78, 2.64) 1.34(0.63, 2.85) 1.60(1.20, 2.15) HbAlc/% 9.95(8.88, 11.28)* 9.90(8.88, 11.90)*# 5.60(5.50, 5.80) TG/(mmol/L) 1.57(1.17, 2.75)* 1.33(0.97, 2.01)*# 1.25(0.92, 1.65) TC/(mmol/L) 5.11±1.02 4.76±1.08 5.02±1.08 HDL-C/(mmol/L) 0.92(0.81, 1.11)* 0.97(0.81, 1.16)* 1.29(1.03, 1.51) LDL-C/(mmol/L) 2.97(2.42, 3.52)* 3.40(2.92, 3.84)*# 2.54(1.98, 3.26) UA/(μmol/L) 330.72±88.18 335.75±106.80 333.67±92.15 FINS/(IU/mL) 8.95(5.90, 11.63) 11.65(8.88, 15.63) 6.5(4.93, 9.00) HOMA-IR 2.71(1.86, 4.16)* 3.76(2.80, 5.93)*# 1.37(1.12, 1.89) Metrnl/(pg/mL) 137.88(131.94, 143.53)* 130.68(126.89, 133.91)*# 142.69(135.13, 152.90) TGF-β/(pg/mL) 131.83(120.60, 157.90)* 120.74(109.46, 134.13)*# 155.29(124.05, 274.47) FBG: 空腹血糖; CRP: C反应蛋白; HbA1c: 糖化血红蛋白; TG: 甘油三酯; TC: 总胆固醇; HDL-C: 高密度脂蛋白胆固醇;
LDL-C: 低密度脂蛋白胆固醇; UA: 血清尿酸; FINS: 空腹胰岛素; Metrnl: 镍纹样蛋白; TGF-β: 转化生长因子-β。
与健康对照组比较, *P < 0.05; 与单纯T2DM组比较, #P < 0.05。表 3 Metrnl和TGF-β含量与一般资料和生化指标的相关性
变量 统计量 Metrnl TGF-β 年龄/岁 r -0.144 -0.197 P 0.071 0.013 性别 r -0.008 0.162 P 0.919 0.042 收缩压/mmHg r -0.385 -0.106 P < 0.001 0.184 舒张压/mmHg r -0.068 0.053 P 0.394 0.505 身高/cm r 0.111 0.081 P 0.164 0.312 体质量/kg r -0.066 -0.080 P 0.412 0.317 体质量指数/(kg/m2) r -0.164 -0.187 P 0.040 0.019 腰围/cm r -0.121 -0.106 P 0.131 0.187 臀围/cm r -0.107 -0.117 P 0.181 0.143 腰臀比 r -0.046 0.019 P 0.566 0.815 病程/年 r -0.494 -0.442 P < 0.001 < 0.001 吸烟 r -0.154 0.059 P 0.053 0.459 FBG/(mmol/L) r -0.313 -0.256 P < 0.001 0.001 CRP/(mg/dL) r 0.050 0.004 P 0.532 0.960 HbAlc/% r -0.402 -0.409 P < 0.001 < 0.001 TG/(mmol/L) r -0.009 0.136 P 0.908 0.089 TC/(mmol/L) r 0.087 0.041 P 0.277 0.611 HDL-C/(mmol/L) r 0.284 0.215 P < 0.001 0.007 LDL-C/(mmol/L) r -0.187 -0.178 P 0.018 0.026 UA/(μmol/L) r -0.025 0.068 P 0.756 0.394 HOMA-IR r -0.292 -0.294 P < 0.001 < 0.001 FINS/(IU/mL) r -0.220 -0.211 P 0.005 0.008 FBG: 空腹血糖; CRP: C反应蛋白; HbA1c:
糖化血红蛋白; TG: 甘油三酯; TC: 总胆固醇;
HDL-C: 高密度脂蛋白胆固醇; LDL-C:
低密度脂蛋白胆固醇; UA: 血清尿酸; FINS: 空腹胰岛素。表 4 糖尿病合并大血管病变多因素Logistic回归分析
变量 偏回归系数 标准误 Wald P OR 95%CI 下限 上限 收缩压 0.096 0.040 5.892 0.015 1.101 1.019 1.190 吸烟 1.929 0.679 8.078 0.004 6.879 1.820 26.009 LDL-C 1.480 0.430 11.867 < 0.001 4.395 1.893 10.204 HOMA-IR 0.410 0.130 9.972 0.002 1.507 1.168 1.945 Metrnl -0.331 0.088 14.258 < 0.001 0.719 0.605 0.853 TGF-β -0.041 0.015 6.922 0.009 0.960 0.932 0.990 LDL-C: 低密度脂蛋白胆固醇; Metrnl: 镍纹样蛋白; TGF-β: 转化生长因子-β。 -
[1] CHEN P Y, QIN L F, LI G X, et al. Endothelial TGF-β signalling drives vascular inflammation and atherosclerosis[J]. Nat Metab, 2019, 1(9): 912-926. doi: 10.1038/s42255-019-0102-3
[2] DADMANESH M, AGHAJANI H, FADAEI R, et al. Lower serum levels of Meteorin-like/Subfatin in patients with coronary artery disease and type 2 diabetes mellitus are negatively associated with insulin resistance and inflammatory cytokines[J]. PLoS One, 2018, 13(9): e0204180. doi: 10.1371/journal.pone.0204180
[3] MIAO Z W, HU W J, LI Z Y, et al. Involvement of the secreted protein Metrnl in human diseases[J]. Acta Pharmacol Sin, 2020, 41(12): 1525-1530. doi: 10.1038/s41401-020-00529-9
[4] 杨婵, 杨嫱妃, 谢子妍, 等. 免疫细胞在糖尿病大血管病变中的作用[J]. 中国比较医学杂志, 2019, 29(3): 98-102. https://www.cnki.com.cn/Article/CJFDTOTAL-ZGDX201903016.htm [5] ELAMRAGY A, YAKOUB S, ABDELGHANY M, et al. Coronary tortuosity relation with carotid intima-media thickness, coronary artery disease risk factors, and diastolic dysfunction: is it a marker of early atherosclerosis?[J]. Egypt Heart J, 2021, 73(1): 34. doi: 10.1186/s43044-021-00157-6
[6] 顾静彧, 吴晓, 张婷, 等. 老年人动脉硬化检测的应用及与血清转化生长因子β1和结缔组织生长因子的相关性研究[J]. 中国全科医学, 2020, 23(5): 529-533. https://www.cnki.com.cn/Article/CJFDTOTAL-QKYX202005025.htm [7] GAO P, WU W, YE J M, et al. Transforming growth factor β1 suppresses proinflammatory gene program independent of its regulation on vascular smooth muscle differentiation and autophagy[J]. Cell Signal, 2018, 50: 160-170. doi: 10.1016/j.cellsig.2018.07.002
[8] 廖占玲, 安国印, 吴晶晶, 等. 冠心病患者血清BMP6、TGF-β1水平与冠状动脉病变程度的相关性[J]. 医学临床研究, 2022, 39(3): 435-438. [9] 牛兆霞, 杨美荣, 牛永菡, 等. 血清Metrnl蛋白水平与2型糖尿病胰岛B细胞功能及胰岛素抵抗的关系[J]. 中国临床研究, 2020, 33(9): 1204-1206, 1210. https://www.cnki.com.cn/Article/CJFDTOTAL-ZGCK202009013.htm [10] 阚春婷, 曹长春. 糖尿病、冠心病患者Metrnl水平与糖脂代谢指标和部分炎性标志物水平的关系[J]. 国际检验医学杂志, 2021, 42(17): 2113-2117. doi: 10.3969/j.issn.1673-4130.2021.17.015 [11] 刘栩晗, 李国生, 李欣宇, 等. 小檗碱抑制TGF-β1通路诱导内脏白色脂肪组织棕色化改善2型糖尿病地鼠脂诱性胰岛素抵抗的研究[J]. 现代中西医结合杂志, 2021, 30(7): 694-699, 713. https://www.cnki.com.cn/Article/CJFDTOTAL-XDJH202107004.htm -
期刊类型引用(2)
1. 于琪力,葛雪,刘丽. 冠状动脉粥样硬化性心脏病患者心力衰竭危险因素分析. 新乡医学院学报. 2024(06): 560-565 .  百度学术
百度学术
2. 孙宁,李飞. 心肌梗死患者血清miR-34a-5p和C1q/肿瘤坏死因子相关蛋白-9水平及其与心肌重构的相关性. 检验医学与临床. 2024(19): 2785-2789 .  百度学术
百度学术
其他类型引用(0)





 下载:
下载:




 苏公网安备 32100302010246号
苏公网安备 32100302010246号